

大家好!欢迎来到今天的科学小课堂。在分子生物学的实验室里,有一种能精准切割 DNA 的 " 基因手术刀",叫限制性内切酶。但 HindⅢ、EcoRI 这些复杂名字总让人摸不着头脑,它们的命名其实藏着一套科学又有趣的规则,今天咱们就来拆解这些 "分子剪刀" 的身份密码。
故事要从 20 世纪 70 年代说起。 1973 年,美国科学家 H.D. Smith 和 D. Nathans 在研究流感嗜血杆菌时,首次提出了酶命名的基本框架,就像给酶发放了第一张 "身份证"。随着基因工程发展,1980 年 R.J. Roberts 完善了这套规则,让全球科学家都能通过统一的名字识别这些重要工具。
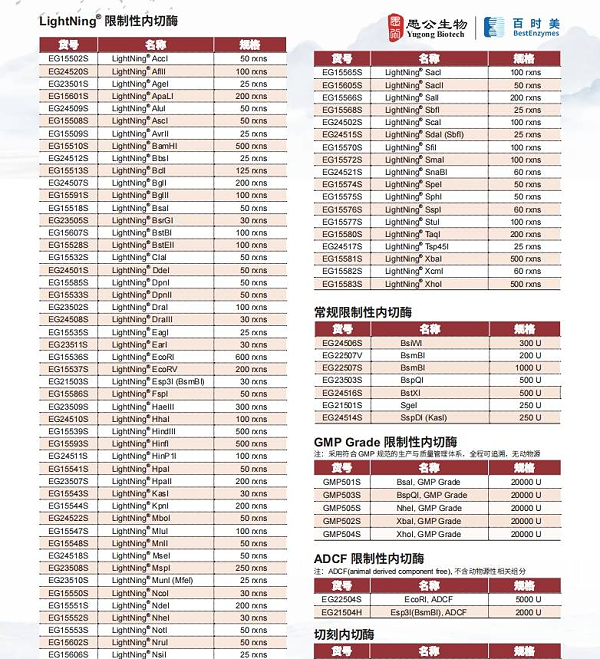
现在咱们来看看命名的关键要素。酶名的前两部分来自微生物的学名:属名首字母大写,比如流感嗜血杆菌属名 Haemophilus 取 "H" ;种名前两个字母小写,influenzae 取 "in",组合成基础代码 "Hin" ,这就像微生物的 "姓名缩写"。第三部分是株系或质粒标识,如果来自流感嗜血杆菌 d 株,就加 "d" 变成 "Hind";要是来自大肠杆菌的 R 质粒,就会有 "R" 这个字母。
第四部分是大写罗马数字,用来区分同一微生物中不同的酶。比如流感嗜血杆菌 d 株先后发现的三种酶,就按顺序叫 HindⅠ 、HindⅡ、HindⅢ,这里的数字只代表发现顺序,和切割功能没关系哦。另外在专业文献里,有时会看到 "R-" 或 "M-" 前缀,"R-" 代表切割 DNA 的限制性内切酶,"M-" 代表给 DNA 穿 " 保护衣" 的修饰酶,比如 R-HindⅢ 和 M-HindⅢ 就是一对 "搭档"。

举个实验室常用的例子 ——EcoRI。它来自大肠杆菌,属名 Escherichia 取 "E",种名 coli 取 "co" ,来自质粒 R 所以加 "R",作为该菌株第一种发现的酶用 "I" ,组合起来就是 EcoRI。另一个例子 BamHI,来自枯草芽孢杆菌 H 株,属名 Bacillus 取 "B",种名 amyloliquefaciens 取 "am",株系 H 加 "H" ,第一种发现用 "I",就成了基因工程中常用的 BamHI。
这套命名体系可不简单,每个字母都藏着信息:看到 EcoRI 就知道来自大肠杆菌,罗马数字帮科研人员快速分辨同菌株的酶,避免实验混淆。更重要的是全球统一的命名打破了语言障碍,让各国科学家能高效沟通,这在基因工程发展中特别重要。
如今基因编辑技术如 CRISPR-Cas9 越来越火,限制性内切酶的应用也在拓展。比如构建基因载体时,正确识别 KpnI 、XhoI 等酶切位点名称是实验成功的关键;分析 DNA 序列时,酶名里的来源信息能帮我们判断是否需要特殊处理甲基化修饰。所以这套诞生于 40 多年前的规则,至今仍是分子生物学的重要基石。
下次再看到 HindⅢ 这样的名字,是不是就像看到一个有故事的 "分子身份证 "?每个字母记录着微生物的来源,数字标注着发现的顺序,背后是科学家探索微观世界的足迹。掌握了这套规则,不仅能解开酶名的密码,还能更懂基因工程的底层逻辑。希望今天的分享能让大家对这些 "基因手术刀 " 多一份亲切,也期待你在科学探索中发现更多有趣的知识。 更多实验室仪器设备和试剂耗材需求请关注苏州阿尔法生物,我们下次再见!